動物発生学研究室
主な研究成果
エピジェネティクス
 脊椎動物のからだを構成する細胞はすべて同じ遺伝情報を持っているが、組織によって様々な個性を持っている。発生初期の未分化な細胞が多様な種類の細胞へと分化していく過程において、クロマチン構造がエピジェネティックに変化することが重要であると考えられているが、実際にどのような構造の変化が分化状態を制御しているのかは未だに明らかになっていない。
脊椎動物のからだを構成する細胞はすべて同じ遺伝情報を持っているが、組織によって様々な個性を持っている。発生初期の未分化な細胞が多様な種類の細胞へと分化していく過程において、クロマチン構造がエピジェネティックに変化することが重要であると考えられているが、実際にどのような構造の変化が分化状態を制御しているのかは未だに明らかになっていない。
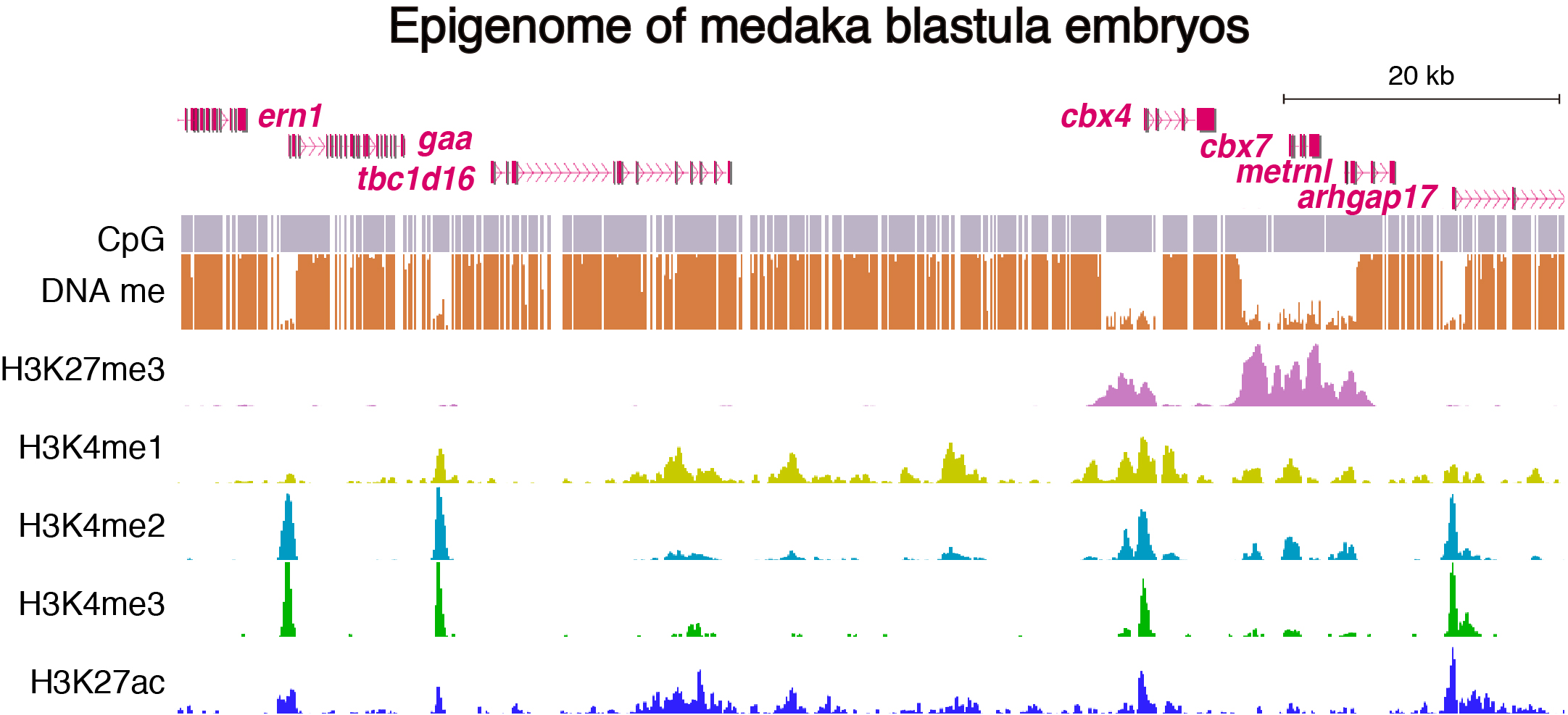
 メダカは、ゲノムサイズが約800Mbと脊椎動物の中では小さく(ヒトの約4分の1)エピゲノム解析に適している。また、分化多能性を持った細胞を発生中の生きた胚から大量に得ることができる。我々はこれらの利点を活かし、メダカの発生初期段階の胚(多分化能を持つ未分化な細胞)と成魚の組織においてDNAおよびヒストンの修飾をゲノムワイドに解析した。その結果、未分化細胞では、発生に特に重要な転写因子をコードする遺伝子座周辺は広範囲にわたりDNA低メチル化状態であることが明らかになった。さらに、この巨大なDNA低メチル化領域の一部は成魚の組織において縮小することを発見し、DNA低メチル化領域のサイズが抑制型のヒストン修飾レベルと相関することを見出した。これによって、未分化細胞においては、分化を制御するような転写因子の発現は巨大なDNA低メチル化領域によって抑制されており、逆に成魚の組織では、低メチル化領域が縮小することで発現が維持されている可能性が示された(Nakamura et al., 2014)。
メダカは、ゲノムサイズが約800Mbと脊椎動物の中では小さく(ヒトの約4分の1)エピゲノム解析に適している。また、分化多能性を持った細胞を発生中の生きた胚から大量に得ることができる。我々はこれらの利点を活かし、メダカの発生初期段階の胚(多分化能を持つ未分化な細胞)と成魚の組織においてDNAおよびヒストンの修飾をゲノムワイドに解析した。その結果、未分化細胞では、発生に特に重要な転写因子をコードする遺伝子座周辺は広範囲にわたりDNA低メチル化状態であることが明らかになった。さらに、この巨大なDNA低メチル化領域の一部は成魚の組織において縮小することを発見し、DNA低メチル化領域のサイズが抑制型のヒストン修飾レベルと相関することを見出した。これによって、未分化細胞においては、分化を制御するような転写因子の発現は巨大なDNA低メチル化領域によって抑制されており、逆に成魚の組織では、低メチル化領域が縮小することで発現が維持されている可能性が示された(Nakamura et al., 2014)。
現在我々は、多分化能を含めた様々な細胞の個性を規定するクロマチン構造の制御メカニズムについて研究を行っている。
- Nakamura, R., Tsukahara, T., Qu, W., Ichikawa, K., Otsuka, T., Ogoshi, K., Saito, T.L., Matsushima, K., Sugano, S., Hashimoto, S., Suzuki, Y., Morishita, S., Takeda, H. Large hypomethylated domains serve as strong repressive machinery for key developmental genes in vertebrates. Development 141, 2568-2580 (2014).
メダカ Da 変異体
 脊椎動物の体幹部で見られる背腹パターンは発生過程において決定される。発生初期における背腹軸形成メカニズムについては知見が多いが、発生後期において背腹のパターンの違いが生まれる過程そのものについてはいまだに不明な点が多い。そこで、発生後期においてメダカ変異体Double anal fin(Da)に着目した。Daは鰭の形態や色素胞の分布など、体幹部の背側表層領域が腹側化するユニークなメダカ自然突然変異体である。最近我々はDa変異体の原因は、新規の巨大なトランスポゾン(Albatross)の挿入によって、zic1およびzic4遺伝子の中胚葉エンハンサーが壊されることにより、体節や尾間充織でその発現が失われることが原因であることを突き止めた(Moriyama et al., 2012)。
脊椎動物の体幹部で見られる背腹パターンは発生過程において決定される。発生初期における背腹軸形成メカニズムについては知見が多いが、発生後期において背腹のパターンの違いが生まれる過程そのものについてはいまだに不明な点が多い。そこで、発生後期においてメダカ変異体Double anal fin(Da)に着目した。Daは鰭の形態や色素胞の分布など、体幹部の背側表層領域が腹側化するユニークなメダカ自然突然変異体である。最近我々はDa変異体の原因は、新規の巨大なトランスポゾン(Albatross)の挿入によって、zic1およびzic4遺伝子の中胚葉エンハンサーが壊されることにより、体節や尾間充織でその発現が失われることが原因であることを突き止めた(Moriyama et al., 2012)。
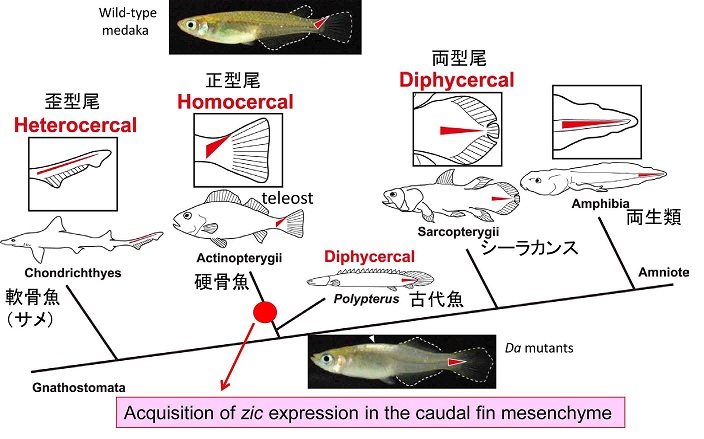 さらに研究の過程で我々は尾部形態の進化とzic遺伝子の関係に注目した。現在、魚類の中で最も繁栄している硬骨魚類の尾部は尾端が背側に屈曲する正型尾となっている。正型尾は真骨魚類に特有の形態であり、水中環境での遊泳に最も適した形態であると考えられている。外見上は背腹対称な形態であるが、内部構造は極端に背腹非対称な形態となっている。尾部骨と呼ばれる脊椎後端部が背側に屈曲し、下尾骨と呼ばれる特殊化した腹側の骨要素が尾鰭を支える形となっている。正型尾は真骨魚類の進化における最も重要な新規形質の一つであると考えられている。野生型メダカは典型的な正型尾を持つが、Da変異体の尾ひれは尾端が屈曲せず、古代魚や四足動物へつながる肉鰭類(シーラカンスなど)の尾の形態(両型尾)に類似している。Da変異体とその原因遺伝子zic1/zic4を手掛かりとし、条鰭類・真骨魚類における正型尾形成機構を明らかした。野生型およびDa変異体の尾部形態の発生とzicの発現の詳細な解析により、硬骨魚類の系譜で尾端の背側間充織でzicが特異的に発現し、その結果背側で骨形成と細胞増殖が抑制されることを見出した。即ち、zicの尾部背側での発現の獲得が硬骨魚類の祖先で起こり、その繁栄に重要なイベントであることが示された(Moriyama et al., 2012)。
さらに研究の過程で我々は尾部形態の進化とzic遺伝子の関係に注目した。現在、魚類の中で最も繁栄している硬骨魚類の尾部は尾端が背側に屈曲する正型尾となっている。正型尾は真骨魚類に特有の形態であり、水中環境での遊泳に最も適した形態であると考えられている。外見上は背腹対称な形態であるが、内部構造は極端に背腹非対称な形態となっている。尾部骨と呼ばれる脊椎後端部が背側に屈曲し、下尾骨と呼ばれる特殊化した腹側の骨要素が尾鰭を支える形となっている。正型尾は真骨魚類の進化における最も重要な新規形質の一つであると考えられている。野生型メダカは典型的な正型尾を持つが、Da変異体の尾ひれは尾端が屈曲せず、古代魚や四足動物へつながる肉鰭類(シーラカンスなど)の尾の形態(両型尾)に類似している。Da変異体とその原因遺伝子zic1/zic4を手掛かりとし、条鰭類・真骨魚類における正型尾形成機構を明らかした。野生型およびDa変異体の尾部形態の発生とzicの発現の詳細な解析により、硬骨魚類の系譜で尾端の背側間充織でzicが特異的に発現し、その結果背側で骨形成と細胞増殖が抑制されることを見出した。即ち、zicの尾部背側での発現の獲得が硬骨魚類の祖先で起こり、その繁栄に重要なイベントであることが示された(Moriyama et al., 2012)。
現在我々は、zicを発現する体節が体幹部背腹パターニングにおいて果たす役割を組織移植で調べるとともに、Zic1/Zic4の標的遺伝子群の単離をマイクロアレイ解析を通じて行っている。
- Kawanishi, T., Kaneko, T., Moriyama, Y., Kinoshita, M., Yokoi, H., Suzuki, T., Shimada, A., Takeda, H. Modular development of the teleost trunk along the dorsoventral axis and zic1/zic4 as selector genes in the dorsal module. Development 140, 1486-96 (2013).
- Moriyama, Y., Kawanishi, T., Nakamura, R., Tsukahara, T., Sumiyama, K., Suster, M.L., Kawakami, K., Toyoda, A., Fujiyama, A., Yasuoka, Y., Nagao, Y., Sawatari, E., Shimizu, A., Wakamatsu, Y., Hibi, M., Taira, M., Okabe, M., Naruse, K., Hashimoto, H., Shimada, A. & Takeda, H. The medaka enhancer mutant for zic1/zic4 provides molecular insights into teleost caudal fin evolution. Curr. Biol. ?22, 1-7 (2012).
ゼブラフィッシュの側線神経回路形成
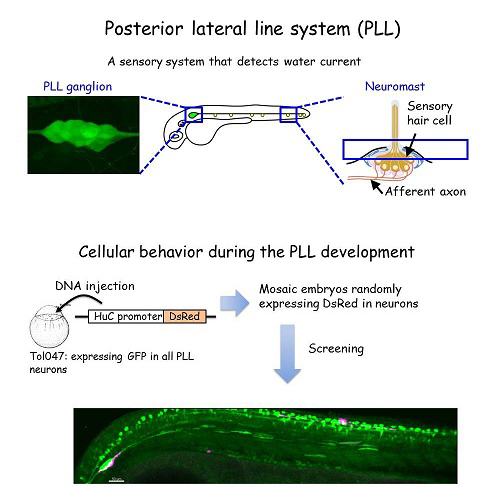 神経回路を構成する各細胞の接続先は細胞レベルで異なるため、均一に見える神経細胞の集団内にも細胞レベルでの細かな差異が見られる。そのため、機能的な回路の形成を理解するには、細胞レベルでの差異に注目し、細胞集団内にその差異を生み出す機構の理解が必要となる。本研究ではこれらの問題に取り組むため、優れたモデル系であるゼブラフィッシュ側線神経系に注目した。この系を用いて、多くの神経系に共通して見られるトポグラフィックマップ;神経細胞-接続先間の対応した位置関係の形成時期とその過程での個々の神経細胞のふるまいを調べた。その結果、初期側線の完成直後(受精2日後)にはトポグラフィックマップが形成されていること、形成過程での神経細胞のふるまい(軸索の伸長速度・成長円錐の形態)は非常に多様であることがわかった。さらに、このようなふるまいの差異がトポグラフィックマップの形成に寄与している可能性が示された(Sato et al., 2010)。
神経回路を構成する各細胞の接続先は細胞レベルで異なるため、均一に見える神経細胞の集団内にも細胞レベルでの細かな差異が見られる。そのため、機能的な回路の形成を理解するには、細胞レベルでの差異に注目し、細胞集団内にその差異を生み出す機構の理解が必要となる。本研究ではこれらの問題に取り組むため、優れたモデル系であるゼブラフィッシュ側線神経系に注目した。この系を用いて、多くの神経系に共通して見られるトポグラフィックマップ;神経細胞-接続先間の対応した位置関係の形成時期とその過程での個々の神経細胞のふるまいを調べた。その結果、初期側線の完成直後(受精2日後)にはトポグラフィックマップが形成されていること、形成過程での神経細胞のふるまい(軸索の伸長速度・成長円錐の形態)は非常に多様であることがわかった。さらに、このようなふるまいの差異がトポグラフィックマップの形成に寄与している可能性が示された(Sato et al., 2010)。
現在我々は側線神経細胞一個ずつの遺伝子発現プロファイルを調べることにより、側線神経節内で神経細胞の個性の違い生み出されるメカニズムを探っている。
- Sato, A., Takeda, H. Neuronal subtypes are specified by the level of neurod expression in the zebrafish lateral line. J Neurosci 33, 556-62 (2013).
- Sato, A., Koshida, S. & Takeda, H. Single-cell analysis of somatotopic map formation in the zebrafish lateral line system. Dev Dyn. 239, 2058-65 (2010).
脊椎動物の左右軸形成機構
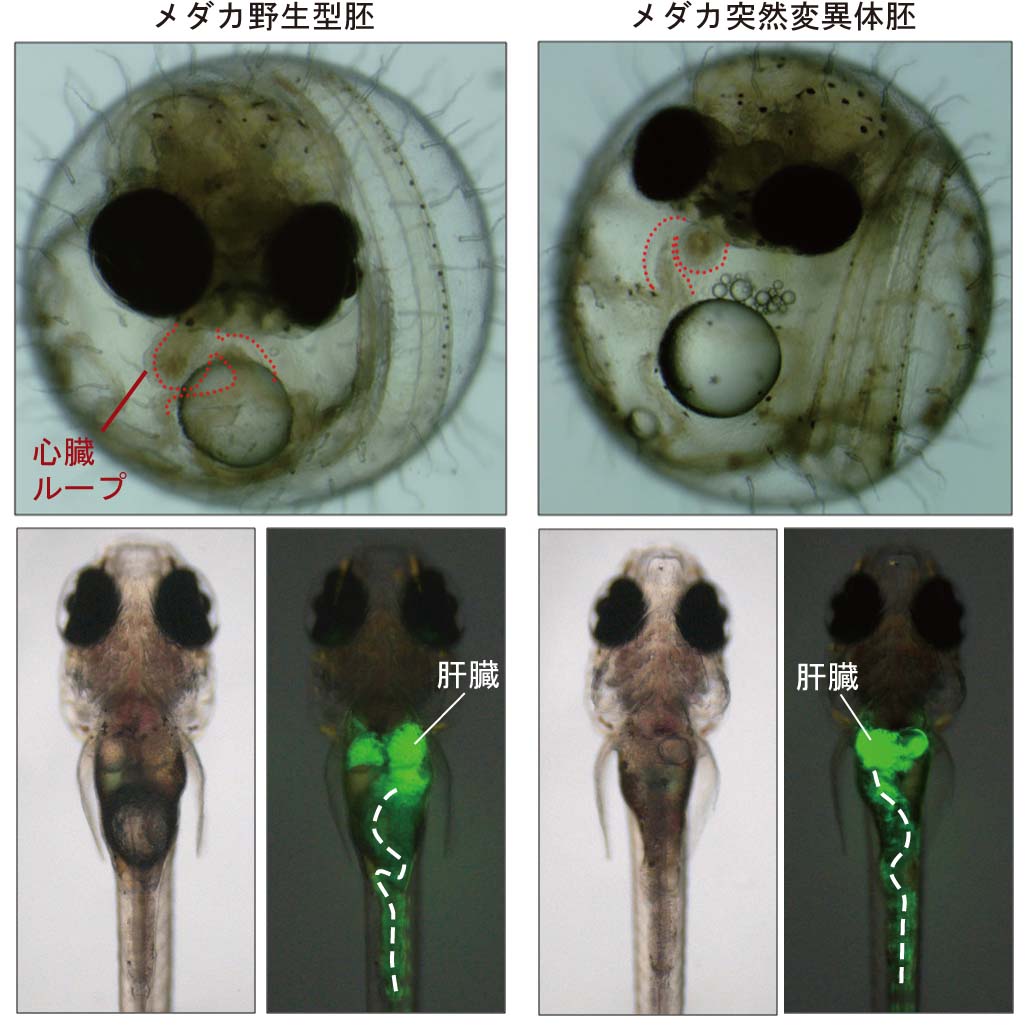 脊椎動物の外形は基本的には左右対称です。しかし、心臓の位置や消化管のねじれ方や肝臓などの配置は明らかに左右非対称となっています。これは狭い空間に多くの器官を配置するための工夫と思われます。重要なことはこれらの非対称性が遺伝的に決まっており個体差がほとんどないということです。つまり私たち人間は、心臓は必ず左にあり、肝臓は右そして大腸は時計回りにまくようになっています。この配置が乱れるのは、人の場合1 : 8,500 - 10,000とされています。この左右非対称性を決めているメカニズムは何でしょう
か。心臓の右側へのルーピングは、脊椎動物胚において共通して最初に現れる外見上の大きな対称性の破れです。母体外で発生し、完全に透明な魚の胚では、心臓は頭部の下に突出しており、外から容易に観察することができます。最近行われたメダカ突然変異体スクリーニングにより、内臓逆位を示すメダカ変異体が合計7系統単離されました。現在これらの突然変異体の原因遺伝子の同定とその機能解析を行っています。
脊椎動物の外形は基本的には左右対称です。しかし、心臓の位置や消化管のねじれ方や肝臓などの配置は明らかに左右非対称となっています。これは狭い空間に多くの器官を配置するための工夫と思われます。重要なことはこれらの非対称性が遺伝的に決まっており個体差がほとんどないということです。つまり私たち人間は、心臓は必ず左にあり、肝臓は右そして大腸は時計回りにまくようになっています。この配置が乱れるのは、人の場合1 : 8,500 - 10,000とされています。この左右非対称性を決めているメカニズムは何でしょう
か。心臓の右側へのルーピングは、脊椎動物胚において共通して最初に現れる外見上の大きな対称性の破れです。母体外で発生し、完全に透明な魚の胚では、心臓は頭部の下に突出しており、外から容易に観察することができます。最近行われたメダカ突然変異体スクリーニングにより、内臓逆位を示すメダカ変異体が合計7系統単離されました。現在これらの突然変異体の原因遺伝子の同定とその機能解析を行っています。
- Kamura, K., Kobayashi, D., Uehara, U., Koshida, S., Iijima, N., Kudo, A., Yokoyama T. & Takeda, H. Pkd1l1 complexes with Pkd2 on motile cilia and functions to establish the left-right axis. Development 138, 1121-9 (2011).
- H. Omran, D. Kobayashi, H. Olbrich, T. Tsukahara, N. T. Loges, H. Hagiwara, Q. Zhang, G. Leblond, E. O’Toole, C. Hara, H. Mizuno, H. Kawano, M. Fliegauf, T. Yagi, S. Koshida, A. Miyawaki, H. Zentgraf, H. Seithe, R. Reinhardt, Y. Watanabe, R. Kamiya, D. R. Mitchell & H. Takeda. Ktu/PF13 is required for cytoplasmic pre-assembly of axonemal dyneins. Nature , 256, 611-616, (2008).
- Hojo, M., Takashima, S., Kobayashi, D., Sumeragi, A., Shimada, A., Tsukahara, T., Yokoi, H., Narita, T., Jindo, T., Kage, T., Kitagawa, T., Kimura, T., Sekimizu, K., Miyake, A., Setiamarga, D., Murakami, R., Tsuda, S., Ooki, S., Kakihara, K., Naruse, K. and Takeda, H. Right-elevated expression of charon is regulated by fluid flow in medaka Kupffer's vesicle. Dev Growth Differ 49, 395-405. (2007).
メダカゲノムの解読とゲノム進化
 日本で開発された実験動物ニホンメダカ(Oryzias latipes, 以下メダカ))は小型魚類としての優れた生物学的特性に加えて、突然変異体コレクションの充実やこれまでの遺伝学的リソースの蓄積により、新しい実験系として注目されている。メダカを用いた研究をさらに加速させることになるのが、今回のゲノム解読である。メダカゲノム解読は情報・システム研究機構 国立遺伝学研究所(遺伝研)と国立大学法人 東京大学(東大)等の共同プロジェクトとして、2002年秋に開始された。本プロジェクトを推進した主な研究室は、遺伝研・小原研究室(配列決定)、東大・新領域・森下研究室(アッセンブルと生物情報)、そして東大・理学系研究科・武田研究室(メダカ生物学・遺伝学)である。
日本で開発された実験動物ニホンメダカ(Oryzias latipes, 以下メダカ))は小型魚類としての優れた生物学的特性に加えて、突然変異体コレクションの充実やこれまでの遺伝学的リソースの蓄積により、新しい実験系として注目されている。メダカを用いた研究をさらに加速させることになるのが、今回のゲノム解読である。メダカゲノム解読は情報・システム研究機構 国立遺伝学研究所(遺伝研)と国立大学法人 東京大学(東大)等の共同プロジェクトとして、2002年秋に開始された。本プロジェクトを推進した主な研究室は、遺伝研・小原研究室(配列決定)、東大・新領域・森下研究室(アッセンブルと生物情報)、そして東大・理学系研究科・武田研究室(メダカ生物学・遺伝学)である。
メダカゲノムはヒトゲノムの4分の1の大きさの約8億塩基と推定されるが、本研究ではその内の約7億の塩基配列を決定し、その中に20,141個の遺伝子を見いだした。今回のメダカゲノム解読は完成度が極めて高く、脊椎動物のゲノム進化について詳細な比較解析により、過去約4億年にわたる魚類ゲノムの大規模な再編成の様子が明らかになった。特に、魚類祖先で起こったゲノムDNA全体が倍加される現象のあと比較的短い期間に大規模再編成が頻繁に起こり、その後メダカでは再編成が抑制されるということがわかった。この発見は今後のゲノム進化の研究におけるメダカの重要性を示唆している。
メダカは日本をはじめとする東アジアに生息するが、性質が異なる様々な地域集団が存在している。今回は主として南日本由来系統の近交系のゲノム解読を行った。さらに、北日本由来系統のゲノムも薄く解読して、DNA塩基配列を比較した。その結果、両者の系統間に約1600万個の違い(1塩基多型(SNP))が見つかった。これはDNA塩基全体の実に3.4%を占め、今まで知られている脊椎動物の種内変異としては最も高い(ヒトは最大でも0.3%程度)。それにもかかわらず、北と南日本由来のメダカは交配可能で、健康な子孫を残すことができる。遺伝子ごとにアミノ酸の変化率を詳しく調べてみると、生殖や性決定に関連する遺伝子群の進化速度は緩やかなことが明らかになった。このことが高い変異率にもかかわらず、両集団間の交配を可能にしていると考えられる。このように、メダカは個別の生物種として分かれる前段階の遺伝的変化を研究するための貴重な材料としても今後期待される。
図の説明
ヒメダカ成魚のペア(雌雄)。このメダカは、今回ゲノム解読が行われた南日本由来の近交系、Hdr-R。
- Sasaki, S., Mello, C. C., Shimada, A., Nakatani, Y., Hashimoto, S., Ogawa, M., Matsushima, K., Gu, S. G., Kasahara, M., Ahsan, B., Sasaki, A., Saito, T., Suzuki, Y., Sugano, S., Kohara, Y., Takeda, H., Fire, A. & Morishita, S. Chromatin-associated periodicity in genetic variation downstream of transcriptional start sites. Science 323, 401-4. (2009).
- Takeda, H. Draft genome of the medaka fish: A comprehensive resource for medaka developmental genetics and vertebrate evolutionary biology. Develop. Growth Differ. 50, S157?S166. (2008).
- Kasahara, M., Naruse, K., Sasaki, S., Nakatani, Y., Qu, W., Ahsan, B., Yamada, T., Nagayasu, Y., Doi, K., Kasai, Y., Jindo, T., Kobayashi, D., Shimada, A., Toyoda, A., Kuroki, Y., Fujiyama, A., Sasaki, T., Shimizu, A., Asakawa, S., Shimizu, N., Hashimoto, S., Yang, J., Lee, Y., Matsushima, K., Sugano, S., Sakaizumi, M., Narita, T., Ohishi, K., Haga, S., Ohta, F., Nomoto, H., Nogata, K., Morishita, T., Endo, T., Shin, I. T., Takeda, H., Morishita, S. and Kohara, Y. The medaka draft genome and insights into vertebrate genome evolution. Nature 447, 714-9. (2007)
- Naruse, K., Hori, H., Shimizu, N., Kohara, Y. and Takeda, H. Medaka genomics: a bridge between mutant phenotype and gene function. Mech Dev 121, 619-28. (2004).
- Kimura, T., Jindo, T., Narita, T., Naruse, K., Kobayashi, D., Shin, I. T., Kitagawa, T., Sakaguchi, T., Mitani, H., Shima, A., Kohara, Y. and Takeda, H. Large-scale isolation of ESTs from medaka embryos and its application to medaka developmental genetics. Mech Dev 121, 915-32. (2004).
メダカ突然変異体を用いた体軸形成、器官形成機構
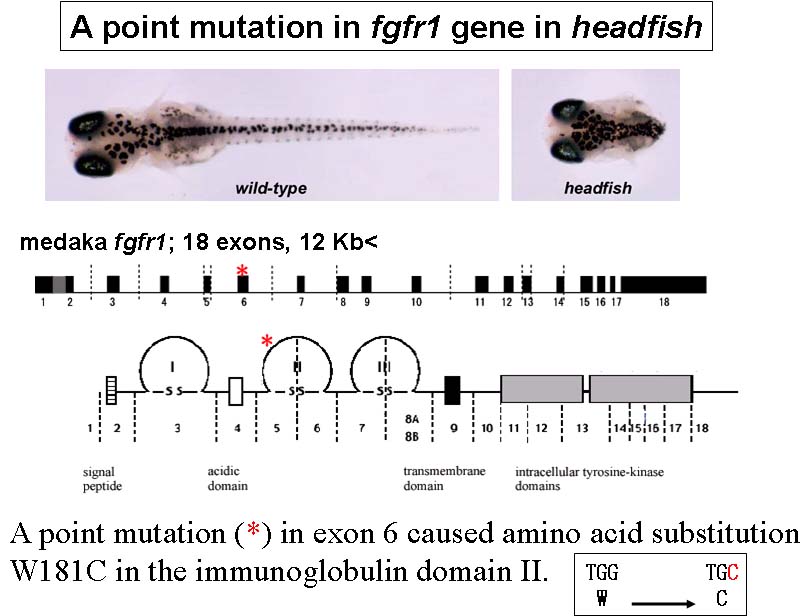 我々は科学技術振興調整費(H12-16, 総括責任者:武田洋幸)の支援によりメダカ突然変異体のスクリーニングを遺伝研、東工大、東大で実施した。得られた変異体のうち、 内胚葉性器官(消化管、肝臓、甲状腺など)、中胚葉性器官(体節、骨、尾芽など)、内臓逆位の変異体に注目して、表現型の解析と原因遺伝子のpositionalクローニングを行っている。また、メダカ自然突然変異体Da, haについても研究を行っている。
我々は科学技術振興調整費(H12-16, 総括責任者:武田洋幸)の支援によりメダカ突然変異体のスクリーニングを遺伝研、東工大、東大で実施した。得られた変異体のうち、 内胚葉性器官(消化管、肝臓、甲状腺など)、中胚葉性器官(体節、骨、尾芽など)、内臓逆位の変異体に注目して、表現型の解析と原因遺伝子のpositionalクローニングを行っている。また、メダカ自然突然変異体Da, haについても研究を行っている。
現在原因遺伝子が判明しているのは以下の変異体である。
tacobo = laminin γ1 (原腸形成、神経管層構造)
headfish = fgfr1 (体幹部欠失)(上図)
ut006 = chordin (頭部欠失)
namako = abcb7 (脂肪肝)
abecobe = uncharacterized (左右軸)
kintoun = novel (左右軸)
aA90 = cytoplasmic dynein2 (左右軸)
- Tsuda, S., Kitagawa, T., Takashima, S., Asakawa, S., Shimizu, N., Mitani, H., Shima, A., Tsutsumi, M., Hori, H., Naruse, K., Ishikawa, Y. & Takeda, H. FAK-mediated extracellular signals are essential for interkinetic nuclear migration and planar divisions in the neuroepithelium. J Cell Sci 123, 484-96. (2010).
- Sano, S., Takashima, S., Niwa, H., Yokoi, H., Shimada, A., Arenz, A., Wittbrodt, J. & Takeda, H. Characterization of teleost Mdga1 using a gene-trap approach in medaka (Oryzias latipes). Genesis 47, 505-513. (2009).
- Miyake, A., Higashijima, S., Kobayashi, D., Narita, T., Jindo, T., Setiamarga, D.,H., E., Ohisa, S., Orihara, N., Hibiya, K., Konno, S., Sakaguchi, S., Horie, K., Imai, Y., Naruse, K., Kudo, A., Takeda, H. A mutation in the abcb7 gene causes abnormal iron and fatty acid metabolism in developing medaka fish. Dev Growth Differ, (2008) in press.
- Shimada, A. and Takeda, H. Production of a maternal-zygotic medaka mutant using hybrid sterility. Dev Growth Differ 50, 421-426. (2008).
- Shimada, A., Yabusaki, M., Niwa, H., Yokoi, H., Hatta, K., Kobayashi, D. and Takeda, H. Maternal-zygotic medaka mutants for fgfr1 reveal its essential role in the migration of the axial mesoderm but not the lateral mesoderm. Development 135, 281-90. (2008).
- Yokoi, H., Shimada, A., Carl, M., Takashima, S., Kobayashi, D., Narita, T., Jindo, T., Kimura, T., Kitagawa, T., Kage, T., Sawada, A., Naruse, K., Asakawa, S., Shimizu, N., Mitani, H., Shima, A., Tsutsumi, M., Hori, H., Wittbrodt, J., Saga, Y., Ishikawa, Y., Araki, K. and Takeda, H. Mutant analyses reveal different functions of fgfr1 in medaka and zebrafish despite conserved ligand-receptor relationships. Dev Biol 304, 326-37. (2007).
- Takashima, S., Shimada, A., Kobayashi, D., Yokoi, H., Narita, T., Jindo, T., Kage, T., Kitagawa, T., Kimura, T., Sekimizu, K., Miyake, A., Setiamarga, D. H., Murakami, R., Tsuda, S., Ooki, S., Kakihara, K., Hojo, M., Naruse, K., Mitani, H., Shima, A., Ishikawa, Y., Araki, K., Saga, Y. and Takeda, H. Phenotypic analysis of a novel chordin mutant in medaka. Dev Dyn 236, 2298-310. (2007).
- Sekimizu, K., Tagawa, M. and Takeda, H. Defective Fin Regeneration in Medaka Fish (Oryzias latipes) with Hypothyroidism. Zoolog Sci 24, 693-9. (2007).
Sasaki, T., Shimizu, A., Ishikawa, S. K., Imai, S., Asakawa, S., Murayama, Y., Khorasani, M. Z., Mitani, H., Furutani-Seiki, M., Kondoh, H., Nanda, I., Schmid, M., Schartl, M., Nonaka, M., Takeda, H., Hori, H., Himmelbauer, H., Shima, A. and Shimizu, N. The DNA sequence of medaka chromosome LG22. Genomics 89, 124-33. (2007).
- Kobayashi, D., Jindo, T., Naruse, K. and Takeda, H. Development of the endoderm and gut in medaka, Oryzias latipes. Dev Growth Differ 48, 283-95. (2006).
- Ohtsuka, M., Kikuchi, N., Yokoi, H., Kinoshita, M., Wakamatsu, Y., Ozato, K., Takeda, H., Inoko, H. and Kimura, M. Possible roles of zic1 and zic4, identified within the medaka Double anal fin (Da) locus, in dorsoventral patterning of the trunk-tail region (related to phenotypes of the Da mutant). Mech Dev 121, 873-82. (2004).
ゼブラフィッシュにおける体節の形成と分節時計
からだの繰り返し構造をつくりだす分節時計
生物は時計を持っている。私たちの神経活動などの日内変動(概日時計)が有名である。一方、時計機構は成体だけでなく、からだ作りの過程(発生)にも重要な役割を持つ。脊椎動物の背骨のような繰り返し構造の創出にはリズムを生み出す時計(分節時計)が存在する。最近我々は、名古屋大学・近藤滋教授(システム生物学)との共同研究により、分節時計の作動原理を解明した。
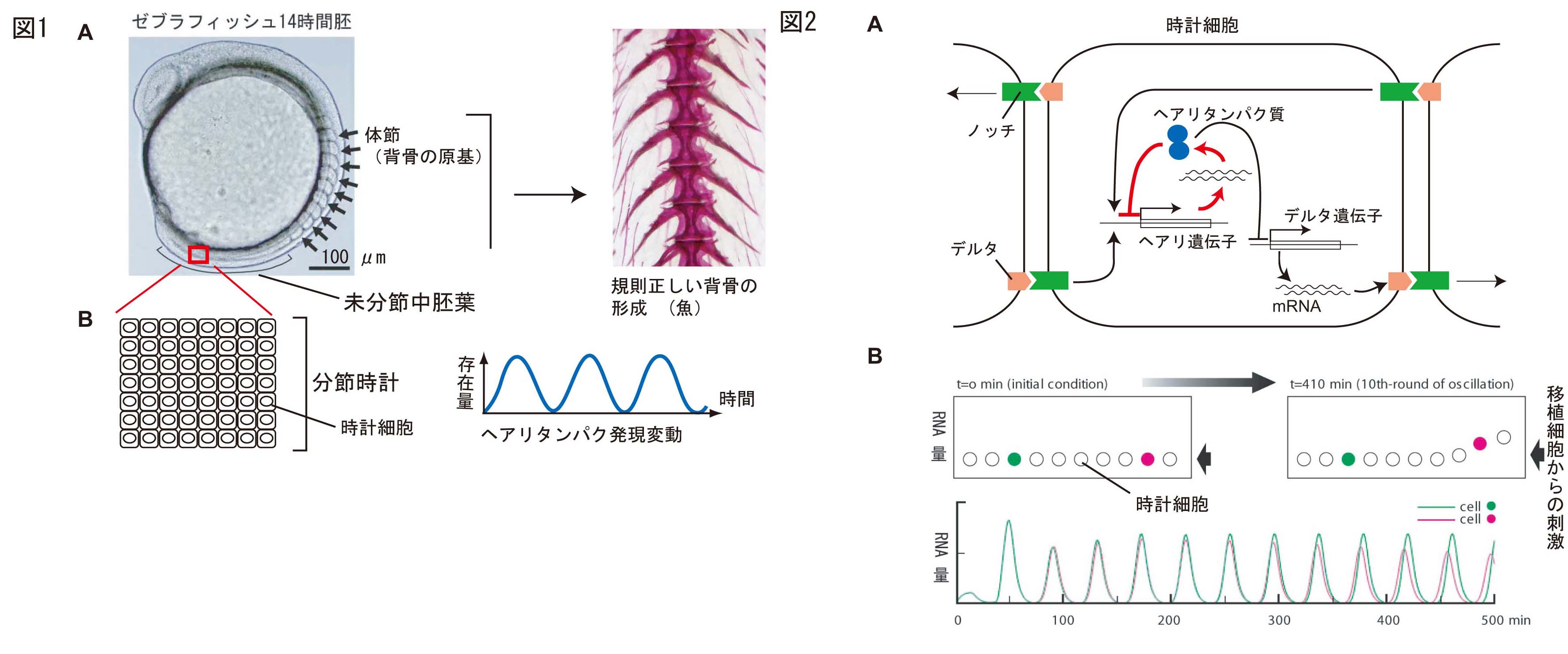
背骨は体節とよばれる中胚葉の細胞塊に由来する。発生中の胚では、体節が前から順番に、中胚葉組織(未分節中胚葉)からくびれ切れる(下図1A)。その様子は、まるで時計で測ったような正確な周期性を持っている(マウスでは2時間、ゼブラフィッシュでは30分毎に一対)。これまでの研究によると、未分節中胚葉に存在する分節時計は数千から数万個の時計細胞の集合体であり、ヘアリとよばれる抑制性転写因子の発現量が周期的に変動することを利用して、時を刻んでいる(図1B)。ヘアリの変動周期は体節形成の周期と一致している。時計細胞では、ヘアリ転写因子を中心としたネガティブ-フィードバックループが形成されており、①転写がON→②ヘアリが一定の濃度に達して、自身の遺伝子の転写がOFF→③転写が止まり、自己分解によりヘアリの濃度が低下する→①・・が一定周期で繰り返している(図2A赤い矢印)。しかし、分節時計が個々の時計細胞を同期させ、正確な時を刻み続ける機構、といったシステムレベルの作動原理はこれまで不明であった。
我々は、細胞間コミュニケーションを担う膜タンパク質、ノッチ(受容体)-デルタ(リガンド)に注目した。図2Aで示された細胞間同期の機構が実際に存在するかどうかを、ゼブラフィッシュ胚を用いた細胞移植実験とシミュレーションを組み合わせて検証した。デルタを恒常的に発現する細胞を正常に振動する分節時計内へ移植すると、移植細胞周辺で時計が加速された。この結果はシミュレーションの予測とよく一致した(図2B)。これらの結果は、ノッチ-デルタを介した位相同期機構が分節時計内に存在していることが示している。さらに詳細な観察の結果、正常に作動している分節時計であっても、細胞分裂や遺伝子発現の不正確さの影響により、時計細胞間の転写タイミングに'ゆらぎ'(ノイズ)がかなり存在していることが判明した。しかしこれらのノイズもまた、ノッチ-デルタを介した位相同期機構により、システムに大きな影響を及ぼさない程度に軽減されていた。このようなノイズ軽減化機構は、ほとんどの遺伝子プログラムにもともと備わっていると考えら、生物のパタン形成の研究に新しい方向性を示したと考えている。
図の説明
図1 A.ゼブラフィッシュにおける体節形成と脊椎骨 B.未分節中胚葉内で機能する分節時計
図2 A.時計細胞内存在する時計機構と細胞間を共役するノッチ-デルタタンパク質
B.時計機構とノッチ-デルタによる細胞間共役を取り込んだシミュレーション:
移植細胞からの刺激による移植細胞周囲の時計が加速されることを示している。事同じ現象が生体内でも観察された。
- Ishimatsu, K., Takamatsu, A. & Takeda, H. Emergence of traveling wave in the zebrafish segmentation clock. Development, 137, 1595-9. (2010).
- Ishimatsu, K., Horikawa, K. and Takeda, H. Coupling cellular oscillators: a mechanism that maintains synchrony against developmental noise in the segmentation clock. Dev Dyn 236, 1416-21. (2007).
- Horikawa, K., Ishimatsu, K., Yoshimoto, E., Kondo, S. and Takeda, H. Noise-resistant and synchronized oscillation of the segmentation clock. Nature 441, 719-23. (2006).
- Terasaki, H., Murakami, R., Yasuhiko, Y., Shin, I. T., Kohara, Y., Saga, Y. and Takeda, H. Transgenic analysis of the medaka mesp-b enhancer in somitogenesis. Dev Growth Differ 48, 153-68. (2006).
- Shimoda, N., Yamakoshi, K., Miyake, A. and Takeda, H. Identification of a gene required for de novo DNA methylation of the zebrafish no tail gene. Dev Dyn 233, 1509-16. (2005).
- Nikaido, M., Kawakami, A., Sawada, A., Furutani-Seiki, M., Takeda, H. and Araki, K. Tbx24, encoding a T-box protein, is mutated in the zebrafish somite-segmentation mutant fused somites. Nat Genet 31, 195-9. (2002).
- Sawada, A., Shinya, M., Jiang, Y. J., Kawakami, A., Kuroiwa, A. and Takeda, H. Fgf/MAPK signalling is a crucial positional cue in somite boundary formation. Development 128, 4873-80. (2001).
- Sakaguchi, T., Kuroiwa, A. and Takeda, H. A novel sox gene, 226D7, acts downstream of Nodal signaling to specify endoderm precursors in zebrafish. Mech Dev 107, 25-38. (2001).
Sakaguchi, T., Kuroiwa, A. and Takeda, H. Expression of zebrafish btg-b, an anti-proliferative cofactor, during early embryogenesis. Mech Dev 104, 113-5. (2001). - Saga, Y. and Takeda, H. The making of the somite: molecular events in vertebrate segmentation. Nat Rev Genet 2, 835-45. (2001).
- Sawada, A., Fritz, A., Jiang, Y. J., Yamamoto, A., Yamasu, K., Kuroiwa, A., Saga, Y. and Takeda, H. Zebrafish Mesp family genes, mesp-a and mesp-b are segmentally expressed in the presomitic mesoderm, and Mesp-b confers the anterior identity to the developing somites. Development 127, 1691-702. (2000).